Meccanismi che modulano la stabilità della forcella di replicazione del DNA e la formazione di R-loops durante i conflitti con la trascrizione.
Progetto
La replicazione e la trascrizione del DNA sono processi che possono interferire l’un l’altro causando danno al DNA, una delle cause principali d’instabilità genomica nei tumori e alte patologie degenerative. Gli scontri frontali tra la replicazione e la trascrizione sono particolarmente deleteri in quanto portano all’accumulo di strutture ricombinogeniche e mutageniche note come R-loops (Brambati et al., 2020).
La DNA/RNA elicasi del lievito S.cerevisiae Sen1 è ortologo della proteina umana Senataxina, la cui funzione è alterata nei tumori e nelle patologie neurodegenerative AOA2/ALS4. Abbiamo dimostrato che Sen1 promuove la progressione e l’integrità della forca di replicazione mentre attraversa i geni molto trascritti (Alzu et al., 2012). In cellule in cui Sen1 è assente, la trascrizione diviene una barriera impenetrabile per la forca di replicazione e specifici meccanismi di salvataggio devono intervenire per impedire la sua rottura e per ultimare la terminazione della replicazione (Brambati et al., 2018).
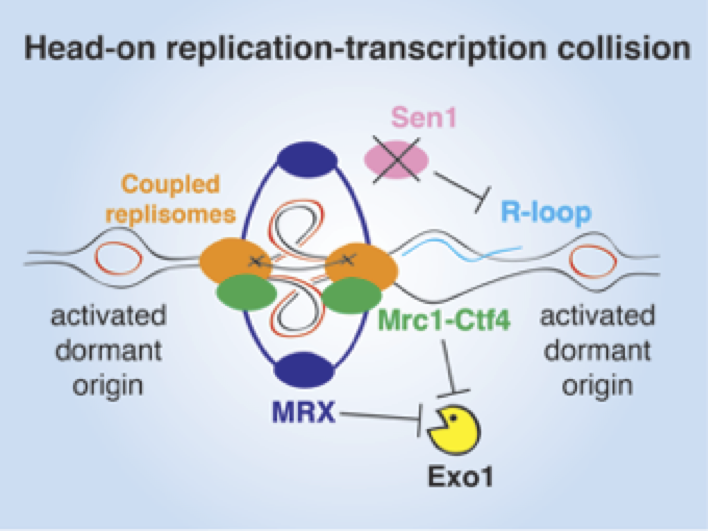
In assenza della DNA/RNA elicasi Sen1 che rimuove gli R-loops, uno scontro frontale tra la replicazione e la trascrizione arresta entrambe le forche generate ad un’origine di replicazione. Nuove forche provenienti da direzioni opposte terminano la replicazione, mentre quelle arrestate sono protette dai complessi MRX e Mrc1-Ctf4 per evitare che vengano processate dalla nucleasi Exo1 (Brambati et al, 2018 NAR).
Il nostro approccio è di utilizzare tecniche genetiche e genomiche, combinate con approcci di biologia molecolare, tra cui l’analisi bi-dimensionale su gel di agarosio degli intermedi di replicazione (Zardoni et al., 2020). Con questi approcci ci proponiamo di esplorare meccanismi e fattori che preservano l’integrità della forca che collide con la trascrizione. Ci proponiamo inoltre di osservare le transizioni patologiche a cui la forcella arrestata dalla trascrizione è soggetta, sia in assenza di Sen1 che di altri fattori che regolano la formazione di R-loops.
Referenze
- Brambati A, Zardoni L, Nardini E, Pellicioli A, and Liberi G (2020). The dark side of RNA:DNA hybrids. Mutat Res 784:108300.
- Alzu A, Bermejo R, Begnis M, Lucca C, Piccini D, Carotenuto W, Saponaro M, Brambati A, Cocito A, Foiani M, and Liberi G. (2012). Senataxin associates with replication forks to protect fork integrity across RNA polymerase II-transcribed genes. Cell 151: 835-846
- Brambati A, Zardoni L, Achar YJ, Piccini D, Galanti L, Colosio A, Foiani M, and Liberi G (2018). Dormant origins and fork protection mechanisms rescue sister fork arrested by transcription. Nucleic Acids Res. 46:1227-1239
- Zardoni L, Nardini E, and Liberi G (2020). 2D gel electrophoresis to detect DNA replication and recombination intermediates in budding yeast. Methods Mol Biol. 2119: 43-59.
